ISME:宏基因组binning基因组揭示了海洋基底奇古菌
出版机构:斯坦福大学地球系统科学系
发??布时间:2020 年 8 月
影响因子:9.18
总结
奇迹是一个丰富而普遍的古菌门,在全球氮和碳循环中发挥着重要作用。最具特征的成员是化学自养氨氧化古菌(AOA ),它们分别解释了土壤和海洋系统中的单细胞生物 5% 和 20% <跨度>。宏基因组测序 分档 获得两个高质量基因组的方法。我们描述了金孢属的一个独特分支,它缺乏氨氧化机制和氨氧化的独特碳固定途径。系统树分析结合了这些基因组。 聚集成海洋门pSL12-like的未培养和大量未研究的pSL12-like分支,呼吸复合物的存在和大量有机碳降解途径证明养分获取的主要方式是有氧异养。两种 MAG 都编码吡咯并喹啉醌 (PQQ ) 依赖性醇脱氢酶,和 III 类型 RuBisCO (核酮糖 -1,5- 二磷酸羧化酶)。代谢重建 暗示存在 RuBisCO 介导的补给 CO2 同化途径可能与中心碳代谢有关。我们认为,这些基因组代表了一种未被认识的进化关系,主要是厌氧的基础物种古细菌与中温带海洋氨氧化古细菌之间的联系,这对研究物种古细菌的多样性具有重要意义。意义。
研究背景
奇异古细菌门是地球上最丰富的微生物,仅占海洋系统单细胞生物的20% , 其成员主要是AOA ,还有一些古菌分支的氨氧化功能尚未得到证实。 pSL12 样 是尚未培养的组,已对 Hypophyta 门的底部分支进行了基因组分析,也称为 1A组 或 阿罗哈 类。虽然一些研究表明 类似pSL12 谱系在氮循环中的作用,但代谢适应性仍有待研究。
实验方法
M1 ( 36.747N , -122.022w , 150m ) 和 M2(36.697N , -122.378w , 200m) 在两个站点采样, 12℃ 培养 8 年, 0.22um 用膜过滤器过滤后 DNA 提取, HiSeq PE150 测序、宏基因组组装 分箱分析 .
实验结果
低多样性宏基因组学中重建的基因组
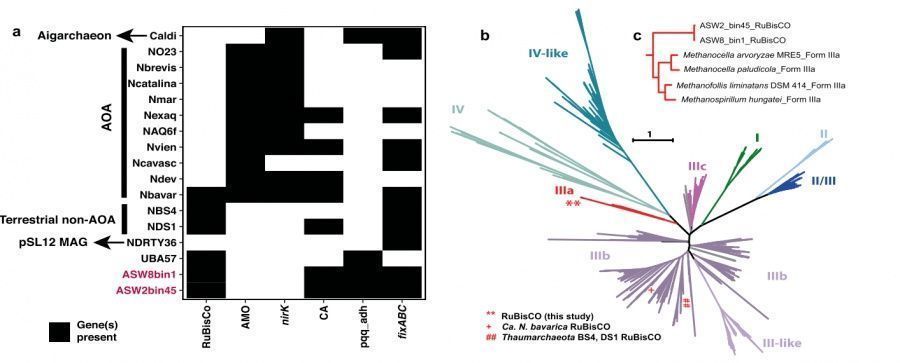
2 样本 ASW2 和 ASW8 进行宏基因组测序和组装 binnig 按框 3 课程 pSL12 谱系基因组,去冗余获取 2 一 MAGs ( ASW2_bin45 , ASW8_bin1 ) ( 完整性分别为 88.8% 和 97.08% ,污染度 <0.3% ( 表格 1)) <跨度>。 11 核心核糖体蛋白级联比较构建最大似然进化树 , 这两个MAGs 位于所有已知的氨氧化分枝杆菌门(海洋 AOA 和土壤 AOA ) 相邻分支(图片 1a ),基于 16S rRNA 基因构建进化树,这两个 MAGS 集群到 pSL12 样 谱系的环境克隆序列分支。
表 1 宏基因组组装的基因组统计

图 1 组装的基因组和类 pSL12 奇异古菌门的一种聚类
与典型海洋奇异古菌的代谢能力差异
两个 MAG 在中未检测到与氨氧化能力相关的信息 AMO (氨单加氧酶)和 nirK 的同源基因不具有奇古菌门特有的固碳途径,也缺少嗜温的AOA。 铜氧化酶的特性与典型的AOA相似 基因组在基因组水平上有很大差异(图 2a <跨度>)。
图
